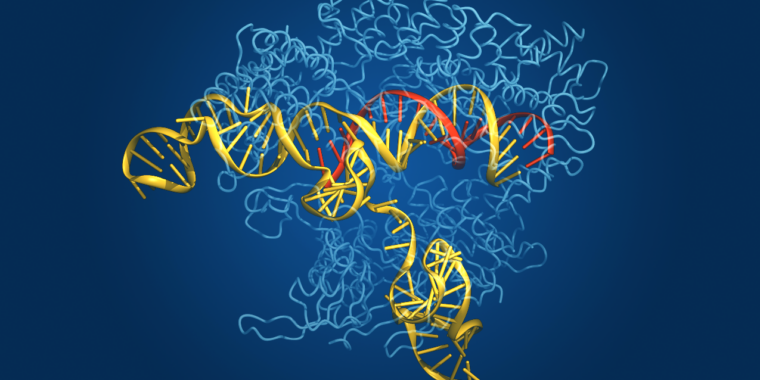
CRISPR — Clustered Often Interspaced Brief Palindromic Repeats — é a resposta do mundo microbiano à imunidade adaptativa. As bactérias não geram anticorpos quando são invadidas por um patógeno e depois mantêm esses anticorpos em suspenso caso encontrem o mesmo patógeno novamente, como nós fazemos. Em vez disso, eles incorporam parte do DNA do patógeno em seu próprio genoma e o ligam a uma enzima que pode usá-lo para reconhecer aquela sequência de DNA patogênico e cortá-la em pedaços se o patógeno aparecer novamente.
A enzima que faz o corte é chamada Cas, de CRISPR associada. Embora o sistema CRISPR-Cas tenha evoluído como um mecanismo de defesa bacteriana, foi aproveitado e adaptado pelos investigadores como uma ferramenta poderosa para manipulação genética em estudos laboratoriais. Também demonstrou usos agrícolas, e a primeira terapia baseada em CRISPR acaba de ser aprovada no Reino Unido para tratar a doença falciforme e a beta-talassemia dependente de transfusão.
Agora, os pesquisadores desenvolveram uma nova maneira de pesquisar genomas de sistemas semelhantes ao CRISPR-Cas. E eles descobriram que podemos ter muitas ferramentas adicionais com as quais trabalhar.
Modificando o DNA
Até o momento, seis tipos de sistemas CRISPR-Cas foram identificados em vários micróbios. Embora difiram nos detalhes, todos têm o mesmo apelo: entregam proteínas a uma determinada sequência de materials genético com um grau de especificidade que até agora period tecnicamente difícil, caro e demorado de alcançar. Qualquer sequência de DNA de interesse pode ser programada no sistema e direcionada.
Os sistemas nativos encontrados nos micróbios geralmente trazem uma nuclease – uma enzima que cliva o DNA – para a sequência, para fragmentar o materials genético de um patógeno. Esta capacidade de cortar qualquer sequência de DNA escolhida pode ser usada para edição genética; em conjunto com outras enzimas e/ou sequências de DNA, pode ser usado para inserir ou excluir sequências curtas adicionais, corrigindo genes mutantes. Alguns sistemas CRISPR-Cas clivam moléculas específicas de RNA em vez de DNA. Estes podem ser usados para eliminar RNA patogênico, como os genomas de alguns vírus, da mesma forma que são eliminados em suas bactérias nativas. Isto também pode ser usado para resgatar defeitos no processamento de RNA.
Mas existem muitas maneiras adicionais de modificar ácidos nucléicos que podem ser úteis. E é uma questão em aberto se as enzimas que realizam modificações adicionais evoluíram. Então, alguns pesquisadores decidiram procurá-los.
Pesquisadores do MIT desenvolveram uma nova ferramenta para detectar matrizes CRISPR variáveis e aplicaram-na a pares de bases de 8,8 tera (1012) de informação genômica procariótica. Muitos dos sistemas encontrados são raros e só apareceram no conjunto de dados nos últimos 10 anos, destacando a importância de continuar a adicionar amostras ambientais que anteriormente eram difíceis de obter a estes repositórios de dados.
A nova ferramenta foi necessária porque as bases de dados de sequências de proteínas e ácidos nucleicos estão a expandir-se a um ritmo ridículo, pelo que as técnicas para analisar todos esses dados precisam de acompanhar. Alguns algoritmos usados para analisá-los tentam comparar cada sequência com qualquer outra, o que é obviamente insustentável quando se trata de bilhões de genes. Outros baseiam-se no agrupamento, mas estes encontram apenas genes que são altamente semelhantes, pelo que não conseguem realmente esclarecer as relações evolutivas entre proteínas distantemente relacionadas. Mas o agrupamento rápido baseado em hashtag, sensível à localidade (“flash clust”) funciona agrupando bilhões de proteínas em menos agrupamentos maiores de sequências que diferem ligeiramente para identificar parentes novos e raros.
A pesquisa usando FLSHclust retirou com sucesso 188 novos sistemas CRISPR-Cas.
Muita CRISPyness
Alguns temas surgiram do trabalho. Uma delas é que alguns dos sistemas CRISPR recentemente identificados usam enzimas Cas com domínios nunca antes vistos, ou parecem ser fusões com genes conhecidos. Os cientistas caracterizaram ainda mais alguns deles e descobriram que um é mais específico do que as enzimas CRISPR atualmente em uso, e outro que corta o RNA que eles propõem é estruturalmente distinto o suficiente para compreender um sétimo tipo inteiramente novo de sistema CRISPR-Cas.
Um corolário deste tema é a ligação de enzimas com diferentes funcionalidades, não apenas nucleases (enzimas que cortam DNA e RNA), com matrizes CRISPR. Os cientistas aproveitaram a notável capacidade de direcionamento genético do CRISPR, ligando-o a outros tipos de enzimas e moléculas, como corantes fluorescentes. Mas a evolução obviamente chegou primeiro.
Como exemplo, FLSHclust identificou algo chamado transposase associada a dois tipos diferentes de sistemas CRISPR. Uma transposase é uma enzima que ajuda um trecho específico do DNA a saltar para outra parte do genoma. A transposição guiada por RNA CRISPR já foi vista antes, mas este é outro exemplo disso. Uma série de proteínas com funções variadas, como proteínas com domínios transmembrana e moléculas de sinalização, foram encontradas ligadas a matrizes CRISPR, destacando a natureza mix-n-match da evolução desses sistemas. Eles até encontraram uma proteína expressa por um vírus que se liga a matrizes CRISPR e as torna inativas – essencialmente, o vírus inativa o sistema CRISPR que evoluiu para proteger contra vírus.
Os pesquisadores não apenas encontraram novas proteínas associadas a matrizes CRISPR, mas também encontraram outras matrizes repetidas regularmente interespaçadas que não estavam associadas a nenhuma enzima cas – semelhante ao CRISPR, mas não ao CRISPR. Eles não têm certeza de qual pode ser a funcionalidade desses sistemas guiados por RNA, mas especulam que eles estão envolvidos na defesa, assim como o CRISPR.
Os autores decidiram encontrar “um catálogo de proteínas guiadas por RNA que expandam nossa compreensão da biologia e evolução desses sistemas e forneçam um ponto de partida para o desenvolvimento de novas biotecnologias”. este trabalho revela flexibilidade organizacional e funcional e modularidade sem precedentes dos sistemas CRISPR”, escrevem. Eles concluem: “Isso representa apenas uma pequena fração dos sistemas descobertos, mas ilumina a vastidão e o potencial inexplorado da biodiversidade da Terra, e o os candidatos restantes servirão como recurso para exploração futura.”
Artigo DOI: 10.1126/science.adi1910